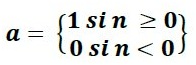La predicción de la duración de la estadía de los
pacientes en la UCI a su ingreso, permite planificar adecuadamente la atención.
Un modelo de estadía basado en múltiples variables de admisión origina
información clínica relevante, como por ejemplo factores de riesgo o patologías
determinantes, y es apropiado para ser tratado por medio de las redes
neuronales artificiales (RNA).
La predicción de la duración de la estadía presenta
problemas que se pueden agrupar en dos categorías: 1) determinación de la
gravedad de la enfermedad de los pacientes que ingresan a la UCI, para lo cual
se utilizan sistemas de índices de gravedad como el APACHE (Acute Physiology
and Chronic Health Evaluation) y APS (Acute Physiology Score), TISS
(Therapeutic Intervention Score System), PSI (Physiologic Stability
Index), que intentan establecer la gravedad de los pacientes en un lapso
que abarca desde el momento de la admisión hasta las 24 h siguientes. Cada uno
de estos índices caracteriza adecuadamente el estado fisiológico del paciente,
aunque se han realizado críticas fundadas respecto de la inclusión de
conocimiento clínico en el momento de la admisión, como se describe en algunas
publicaciones, y 2) una gran cantidad de trabajos intentan predecir estadía,
usando métodos estadísticos lineales multivariados. Estos estudios adolecen de una extrema simplificación del
modelo, que exige variables no correlacionadas, limitando el número de
variables y no permitiendo el tratamiento simultáneo de diferentes tipos de
variables (nominales, continuas o binarias). Estos modelos tampoco consideran
las complejas interacciones que existen entre las diferentes variables. Las
dificultades descritas aumentan en el caso de una UCI, por la diversidad de la
patología y por el compromiso de múltiples órganos y sistemas.
Recientemente se han publicado estudios que utilizaron
modelos no lineales para predecir estadías, como las RNA, pero que están
circunscritos a aplicaciones particulares y de este modo no satisfacen las exigencias necesarias para
el modelado de los casos que requieren las prestaciones de una UCI.
Una red
neuronal artificial es un modelo computacional compuesto de elementos
matemáticos que se han diseñado para realizar aproximadamente el trabajo que
efectúan las neuronas. Está constituida por una capa que recibe y organiza las
entradas, una capa de "neuronas" intermedia y una capa de
"neuronas" de salida, unidas por conexiones que representan las
sinapsis neuronales. Los patrones particulares son representados por los
valores de las conexiones. Las redes neurales pueden ser entrenadas por un
método de cálculo de conexiones denominado "back-propagation",
que es una extensión del método de los mínimos cuadrados utilizado en la
regresión lineal.
En el
presente estudio se utilizaron las variables más relevantes del índice APACHE
II y se cuantificó la información
clínica de admisión, usando las hipótesis diagnósticas para conformar un nuevo
factor (Diagnósticos de Ingreso), que representó la situación de los pacientes
al ingreso, lo que condujo a un proceso de enriquecimiento de los datos por la adición de información
clínica de expertos, que permitió cuantificar los diagnósticos asociados a cada
paciente. Estos procedimientos llevaron a una mejor cobertura de la información
requerida para la predicción de la estadía.
Para
optimizar el modelo se utilizó el paradigma de RNA multicapa y el algoritmo de
aprendizaje "Back propagation", que permitió utilizar diferentes
tipos de variables y representar adecuadamente las no-linealidades y
sinergismos de las condiciones de ingreso de los pacientes.
MATERIAL
Y MÉTODO
Recolección
de datos: Los datos fueron obtenidos de pacientes de dos
UCI para adultos de la ciudad de Santiago: 1) UCI del Hospital Barros Luco
Trudeau (HBLT), que realiza en promedio 350 internaciones anuales, y que aportó
162 casos a este estudio; 2) UCI del Hospital de Carabineros, que realiza 150
internaciones anuales en promedio, y que aportó 132 casos. La muestra obtenida
constó de 294 casos (243 sobrevivientes y 51 fallecidos), con un rango de
estadía que varió entre 1 y 42 días.
Las
variables estudiadas en cada paciente se agruparon en cuatro factores:
Epidemiológico, Condición de gravedad, Indice fisiológico (APS) y Diagnósticos
de ingreso. Cada uno de estos factores estuvo compuesto a su vez por variables
individuales, alcanzando un total de 36 variables de entrada (Tabla 1). Los datos fueron recolectados retrospectivamente
de los registros clínicos de cada unidad, por alumnos del quinto año de la
Carrera de Medicina de la Facultad de Ciencias Médicas de la Universidad de
Santiago de Chile y enfermeras de la UCI del HBLT. La mayoría de las variables
pudo ser cuantificada directamente pues fueron binarias o continuas. La
incorporación del factor Diagnóstico de ingreso originó un problema de
cuantificación, ya que se debió incluir conocimiento médico para convertirlo en
variables numéricas tratables por la red neuronal.
Cuantificación
de los diagnósticos. El procedimiento adoptado para
cuantificar los diagnósticos consistió de cuatro pasos:
1)
Se dividieron los 1.200 diagnósticos (para cada paciente se consideró un
diagnóstico principal y hasta 7 estados co-mórbidos de relevancia) en 17 grupos
que representaron sistemas fisiológicos y grupos mórbidos (Tabla 2).
2)
A continuación, los especialistas clasificaron cada diagnóstico en tres
diferentes categorías: crónico, agudo e hiperagudo (excepto el caso de los
diagnósticos de trauma, que fueron considerados todos agudos o hiperagudos).
3)
En el tercer paso, se debió tener en cuenta que en cada sistema fisiológico o
grupo mórbido podía existir más de una de estas categorías, o existir una
combinación de éstas y se generó un sistema de clasificación que tenía asociado
un orden creciente de gravedad, como se muestra en la Tabla 3.
4)
Finalmente, se desarrolló un algoritmo que asignó un valor de gravedad a cada
uno de los 17 grupos para cada paciente según la combinación de diagnóstico
obtenida. Así, cada paciente quedó representado por 36 variables numéricas que
conformaron los patrones de entrada ingresados a la red neuronal.
Modelado.
Las RNA son representaciones lógicas altamente simplificadas de grandes grupos
de neuronas simuladas en "software". Estos modelos no requieren
información a priori, y obtienen la información exclusivamente
desde un conjunto de datos. El fundamento de estos modelos se basa en el
funcionamiento de las redes de neuronas del hipocampo17,19, donde la información se almacena en
las sinapsis neuronales. De este modo, en una estructura de red previamente
fijada, para desarrollar el modelo sólo se requiere un método de cálculo
(denominado aprendizaje) que permite obtener los valores adecuados de las
sinapsis que logren un error mínimo entre las salidas del modelo y los datos de
salida resultantes del fenómeno real. Desde un punto de vista funcional, estos
modelos son similares a una regresión estadística, la cual relaciona variables
independientes (entradas) con una variable dependiente (salida), mediante una
función lineal. La ventaja de una RNA del tipo hetero-asociativa (que relaciona
variables de entrada y salida con una o más capas de neuronas intermedias,
denominadas capas ocultas) sobre una regresión, es que permite encontrar
cualquier tipo de relación entre las variables, proceso que es frecuente en la
práctica clínica. El modelo utilizado en este trabajo estuvo formado por una
red hetero-asociativa con una capa intermedia, como se muestra en la Figura 1. El método utilizado para obtener los
valores de las sinapsis (representadas por las conexiones en la Figura 1) fue el "Back propagation" que permite a la red aprender
efectivamente. Este algoritmo se basa en una aplicación iterativa del método de
los mínimos cuadrados usado en el cálculo de regresión. El aprendizaje (cálculo
iterativo) se basa en la presentación simultánea de los datos de entrada y los
datos de salida a la red, para todo el conjunto de casos (ciclo de
entrenamiento). El principal problema del aprendizaje es lograr disminuir el
error de salida sin introducir sesgo, para lo cual se requiere evaluar el error
en un conjunto diferente de casos (conjunto de prueba) que no fueron usados en
la etapa de aprendizaje. Durante el aprendizaje se debe ajustar básicamente dos
parámetros: la tasa de aprendizaje (< 1, correspondiente al paso del método
del gradiente) y el número de neuronas de la
capa oculta. Un método adecuado para eliminar el sesgo del modelo y obtener el
número adecuado de neuronas en la capa intermedia, es usar el método de la
validación cruzada el cual consiste en separar el
conjunto de datos en varios grupos, dejando uno para prueba y el resto para
entrenamiento. Posteriormente se cambia el grupo de prueba y se continúa hasta
probar todos los grupos. Después de lograr una red debidamente entrenada, es
posible obtener la importancia o el impacto de las variables de entrada sobre
la salida (en este caso la estadía). Para lograr este fin, se requiere realizar
un análisis de sensibilidad de la red, el cual consiste en producir variaciones
en las entradas y observar los cambios en las salidas. Así, la entrada que
produzca los mayores cambios en la salida será aquella que cause un mayor
impacto en la estadía.
Figura 1. Modelo de la Red Neuronal Artificial
para estimar estadía, con cuatro factores de entrada (36 variables), una capa
oculta y la estadía como salida.
|
RESULTADOS
En
nuestro estudio, para el entrenamiento de redes que utilizaron 36 variables de
entrada, los modelos desarrollados emplearon de 10 a 60 neuronas en la capa
oculta. Los mejores resultados se obtuvieron con 45 neuronas en la capa oculta,
con una tasa de aprendizaje de 0,5 en la capa oculta; 0,1 en la capa de salida,
realizándose 2.714 ciclos de entrenamiento, con lo que se obtuvo un error de
11% (4,5 días).
La
evaluación de los errores individuales demostró que los errores mayores se
producen en las estadías largas (> 30 días), mientras que los casos en los
cuales la estadía es menor que 30 días el error promedio es de 8,7% (3,56 días
de promedio). La variación en este caso resultó ser de ± 0,4 días (IC 95%) y el
coeficiente de correlación entre las estadías reales y los valores predichos
por el modelo es r=0,90 (p <0,001). El análisis de sensibilidad permitió
calcular el porcentaje de importancia que tuvo cada variable en predecir la
estadía. Estos resultados se presentan en la Tabla 4, que indica el porcentaje que
corresponde a cada factor en el que se agrupan las variables.
Con el
fin de examinar más detalladamente la selección de variables se desarrolló un
nuevo modelo, que incluyó las 12 variables mostradas en la Tabla 4. Cuando se utilizó una arquitectura de
29 neuronas en la capa oculta, con una tasa de aprendizaje de 0,5 y en 2.203
ciclos de entrenamiento, se logró un error de 8,8% (3,6 ± 0,4 días, IC 95%). El
análisis de sensibilidad en este modelo reducido estableció un orden similar de
las variables mostradas en la Tabla 4, sólo descendió en importancia la
variable "Infección al ingreso", mientras se hizo más relevante la
variable "Frecuencia respiratoria".
DISCUSIÓN
Para
evaluar el poder de predicción del modelo se puede realizar una comparación
simple entre los valores predichos por la red y la predicción realizada por el
valor promedio de las estadías, para un diagnóstico particular. Las
predicciones de estadía obtenidas utilizando redes neuronales son mejores que
aquellas obtenidas por un sistema de predicción por promedio, como se muestra
en la Tabla 5, al comparar los errores promedios para
el grupo de diagnósticos seleccionados con el análisis de sensibilidad.
Una
medida adecuada de comparación entre diferentes modelos es el uso del
coeficiente de correlación. Una selección de trabajos recientes de predicción
de estadía en UCI, mediante modelos lineales, permite destacar los trabajos de
Clark et al y
Chan et al que muestran coeficientes de correlación de
r=0,76 (con 2.672 pacientes) y r=0,85 (con 1.064 pacientes), al usar variables
del índice APACHE como entradas.
Entre
los trabajos que usan RN para predecir estadía se puede destacar la comparación
con modelos lineales que realiza Zernikow et al para 2.144 pacientes admitidos
en unidades de neonatología, donde los modelos lineales logran correlaciones de
r=0,85 y las RN de r=0,87. Se debe hacer notar que para los elevados valores de
r y número de casos que se presentan en la literatura, todos los coeficientes
de correlación mencionados son significativos con valores p <0,001.
Los
errores obtenidos con conjuntos independientes de prueba fueron relativamente
bajos para la predicción de 30 días (3,56 días), dado que pudo existir un error
no previsible en los datos por razones netamente administrativas, ya que en
numerosas ocasiones se puede retrasar la salida de los pacientes hasta por dos
días. El origen más probable para el aumento de los errores cuando la estadía
superó los 30 días, fue la falta de información en algunas variables de entrada
y no la capacidad del modelo, dado que los resultados con el modelo reducido (12
variables) tuvieron errores similares a aquellos obtenidos con el modelo
general (36 variables). Problemas similares han sido descritos por otros
autores.
Desde
el punto de vista de la evolución clínica, los pacientes con permanencias
mayores a 30 días, fueron influidos en forma importante por las eventuales
complicaciones que se produjeron al interior de la UCI. Para caracterizar estos
cambios, proponemos usar en el futuro índices que evalúen estados de evolución,
los cuales adquieren información durante la estada del paciente.
El
análisis de sensibilidad apoyó la propuesta inicial de incluir las hipótesis
diagnósticas, ya que el factor "Diagnósticos de ingreso" resultó
responsable del 51,65% de la variación de la estadía. Este hallazgo solucionó
uno de los principales problemas por el cual son criticados los índices de
gravedad, la exclusión del conocimiento clínico al ingreso del paciente.
El
proceso de cuantificación diagnóstica es difícil y requiere del aporte
fundamental de los especialistas, que garanticen su reproducibilidad en
cualquier especialidad médica. El presente estudio demostró que los
diagnósticos de ingreso priman en importancia predictiva sobre otras variables
cuantitativas, como los parámetros fisiológicos.
Como un
hecho relativamente sorprendente, las patologías de los sistemas digestivo,
respiratorio y renal son responsables de una parte importante de la
prolongación de la estadía, primando sobre las patologías neurológicas y
cardíacas, que no aparecen en nuestro estudio como determinantes de la
prolongación de la estadía.
La
inclusión de patologías psiquiátricas resultó adecuada, ya que se incluyen en
ellas los diagnósticos de alcoholismo, que en general presentan permanencia
prolongada.
En base
al presente estudio, las perspectivas de desarrollo a futuro de las UCI
debieran incluir la utilización de sistemas computacionales que recojan
información al ingreso y efectúen predicciones en línea al momento de la
admisión. También debiera considerarse la incorporación de los costos de las prestaciones
en la unidad y estudiar su relación con la estadía.